Forschungsschwerpunkte
DrugDiff

Unsere Forschungsgruppe hat DrugDiff entwickelt, ein latentes Diffusionsmodell für die Erzeugung kleiner wirkstoffähnlicher Moleküle, das sich an den Fortschritten im Bereich der KI-gestützten Bildgenerierung orientiert. Durch die Einbeziehung von Steuerungsmöglichkeiten in den Generierungsprozess können die Nutzer bestimmte molekulare Eigenschaften der erzeugten Verbindungen wirksam beeinflussen. Unsere Experimente haben beispielsweise gezeigt, dass sich Eigenschaften wie logP, Synthetisierbarkeit und Arzneimittelähnlichkeit gezielt steuern lassen. Durch die Modulation der „Steuerungsstärke“ beobachteten wir erkennbare Verschiebungen in den Eigenschaftsverteilungen der erzeugten Moleküle, und unsere Ergebnisse zeigen eine gute Generalisierung über chemisch unterschiedliche kleine Moleküldatensätze. Wir erforschen derzeit das volle Potenzial der anpassungsfähigen Architektur von DrugDiff, einschließlich seiner Modularität und Eigenschaftsagnostik, um die kontrollierte Molekülgenerierung weiter zu verbessern
PriSyn
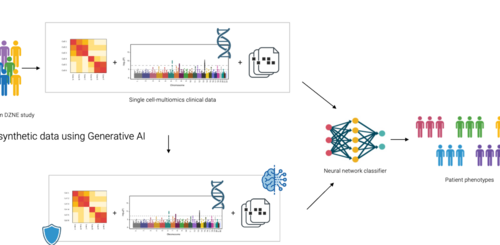
Das PriSyn-Projekt ist eine kollaborative Initiative, die sich auf die Umgestaltung der Nutzung von Gesundheitsdaten für Forschungszwecke konzentriert. Während medizinische Daten wie genetische Informationen und Blutwerte eine wichtige Bedeutung haben, stellen die strengen Beschränkungen und der kontrollierte Zugang zu diesen Daten eine Herausforderung für die gemeinsame Nutzung und die Verwendbarkeit von Daten aus verschiedenen Quellen dar. Die wichtigsten Partner in diesem Projekt sind das Deutsche Zentrum für Neurodegenerative Erkrankungen (DZNE), das CISPA Helmholtz-Zentrum für Informationssicherheit, das Start-up QuantPi und das IT-Unternehmen Hewlett Packard Enterprise (HPE). Ihr Hauptaugenmerk liegt auf der effizienten und datenschutzgerechten Nutzung medizinischer Daten unter Wahrung des Datenschutzes. Das Projekt schlägt dabei eine innovative Methode zur Anonymisierung von Daten vor, die im Gegensatz zu früheren Methoden, bei denen den Daten Rauschen hinzugefügt wurde, maschinelle Lernmodelle verwendet, die unter dem Mechanismus der differentiellen Privatsphäre trainiert werden, um künstliche (synthetische) Daten zu erzeugen. Die erzeugten synthetischen Daten spiegeln die statistischen Eigenschaften der realen Daten wider und gewährleisten gleichzeitig eine breitere Zugänglichkeit sowie die Wahrung der Privatsphäre.
CISPA wird die Schlüsselrolle bei der Erzeugung der künstlichen Daten spielen. Das DZNE wird sich auf den Aufbau eines multizentrischen Einzelzelldatensatzes konzentrieren, der klinische Metadaten, Einzelzelltranskriptomdaten und Genotypen enthält. Darüber hinaus werden sie an der Erstellung eines neurodegenerativen Klassifikators beteiligt sein und als Teil der Vorverarbeitung biologisch plausible Metriken erstellen, um die mit den synthetischen Daten erstellten Modelle zu bewerten. QuantPi wird eine entscheidende Rolle bei der Bewertung der Qualität der synthetischen Daten für verschiedene Anwendungsfälle spielen. Dazu gehört die Identifizierung von Messgrößen und Benchmark-Experimenten, mit denen der Kompromiss zwischen Datenschutz und Datennutzen genau analysiert werden kann. HPE wird dafür verantwortlich sein, dass die im Rahmen des Projekts entwickelten generativen Modelle effizient in benutzerfreundliche und sichere Hardwarelösungen integriert werden.
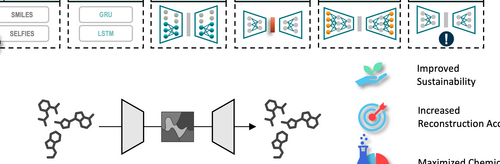
Small Molecule Autoencoders
Autoencoder werden häufig eingesetzt, um Einbettungen für molekulare Daten zu erstellen, die in nachfolgenden Deep-Learning-Anwendungen verwendet werden. Es gibt jedoch eine bemerkenswerte Lücke bei der Bewertung der Qualität der chemischen Informationen, die in den latenten Räumen erfasst werden, und die Auswahl der Modellarchitekturen scheint oft zufällig zu sein. Solche nicht optimierten Architekturen können die Integrität des latenten Raums beeinträchtigen und zu einem übermäßigen Energieverbrauch während des Trainings führen, wodurch die Modelle weniger nachhaltig sind. Wir haben eine Reihe von systematischen Experimenten durchgeführt, um die Auswirkungen der Autoencoder-Architektur auf die Genauigkeit der molekularen Rekonstruktion und die Qualität des latenten Raums zu untersuchen. Unsere Ergebnisse zeigen, dass wir durch die Optimierung der Architektur die gleiche Leistung wie bei generischen Architekturen erreichen können, während wir 97 % weniger Daten verwenden und den Energieverbrauch um etwa 36 % senken. Darüber hinaus haben unsere Experimente gezeigt, dass die Verwendung von SELFIES für die molekulare Repräsentation zu einer Verringerung der Rekonstruktionsgenauigkeit im Vergleich zu SMILES-Repräsentationen führt. Außerdem führte das Training mit enumerierten SMILES zu einer signifikanten Verbesserung der Qualität des latenten Raums.
PRO-GENE-GEN
Genetische Daten sind hochsensible Informationen, die durch strenge gesetzliche Vorschriften geschützt sind, was ihre Weitergabe erschwert. Die Nutzung genetischer Informationen birgt jedoch ein großes Potenzial für die Diagnose und Behandlung von Krankheiten und ist für die Verwirklichung der personalisierten Medizin unerlässlich. Es wurden zwar Mechanismen zur Wahrung der Privatsphäre entwickelt, jedoch sind diese entweder mit erheblichem Aufwand verbunden oder können die Privatsphäre sensibler Patientendaten nicht vollständig schützen. Dies schränkt die Möglichkeit ein, Daten mit der Forschungsgemeinschaft zu teilen, was die wissenschaftliche Entdeckung und die Reproduzierbarkeit der Ergebnisse behindert. Daher schlagen wir einen anderen Ansatz vor, bei dem synthetische Datensätze verwendet werden, die die Eigenschaften von Patientendatensätzen teilen und gleichzeitig die Privatsphäre respektieren. Wir erreichen dies, indem wir die neuesten Fortschritte in der generativen Modellierung nutzen, um virtuelle Kohorten zu synthetisieren. Solche synthetischen Daten können mit etablierten Toolketten analysiert werden, der wiederholte Zugriff beeinträchtigt nicht das Budget für die Privatsphäre und kann sogar offen mit der Forschungsgemeinschaft geteilt werden. Während die generative Modellierung hochdimensionaler Daten, wie z. B. genetischer Daten, bisher unerschwinglich war, haben die jüngsten Entwicklungen im Bereich der tiefen generativen Modelle eine Reihe von Erfolgsgeschichten in einer Vielzahl von Bereichen gezeigt. Die Ergebnisse des Projekts sind in unserem Papier zusammengefasst, das auf der PoPETS 2024 angenommen wurde.
Swarm Learning
Wir sind Teil der Swarm Learning-Aktivitäten am DZNE und arbeiten an mehreren Projekten im Zusammenhang mit hochdimensionaler Durchflusszytometrie auf globaler Ebene, medizinischer Bildgebung und der gemeinsamen Feinabstimmung großer Sprachmodelle. Darüber hinaus untersuchen wir den Einsatz von Schwarmlernen in generativen Modellen.
Modular HPC
Wir nutzen HPC- und GPU-Cluster am DZNE, entwickeln Tools und Pipelines und arbeiten gemeinsam mit der DZNE-Community an der Übertragung von Rechenaufgaben auf unsere Cluster mithilfe von Containerisierung, wobei wir uns an Best Practices für Reproduzierbarkeit und Forschungssoftwaretechnik halten. Auf der Grundlage unseres starken informatischen Hintergrunds evaluieren wir kontinuierlich neue Technologien wie FPGAs (Field Programmable Gate Arrays), die die Energieeffizienz beim maschinellen Lernen und anderen Aufgaben auf die nächste Stufe heben können, und arbeiten an deren Integration in DZNE-Forschungsprojekte.